前几天有同学问了一篇文章里的一个方法的实现,看了一下这篇文章除了
qPCR验证基本都是纯生信,今天就试着来复现一下。随缘复现哈,如果阅读数据不好看的话,可能就放弃了,希望大家多多点赞、在看,转发支持。

文章标题:Investigation of a Hypoxia-Immune-Related Microenvironment Gene Signature and Prediction Model for Idiopathic Pulmonary Fibrosis.
doi: 10.3389/fimmu.2021.629854
注:因为只是浅复现一下,个人水平也有限,过程中可能曲解作者的原意,难免存在纰漏或瑕疵,希望大家批评指正。
流程
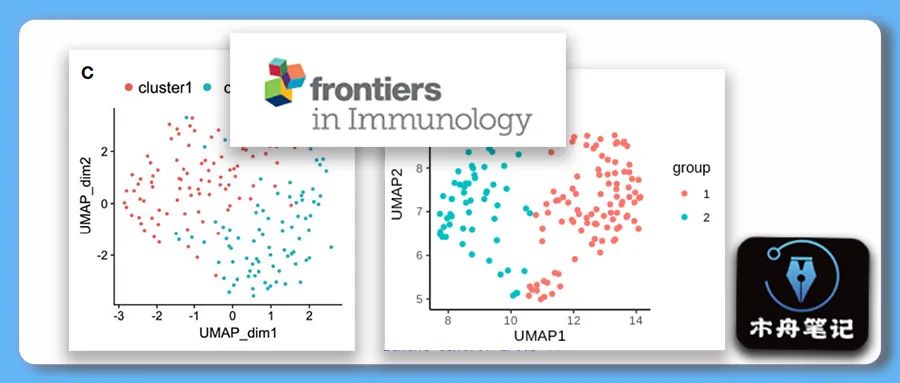
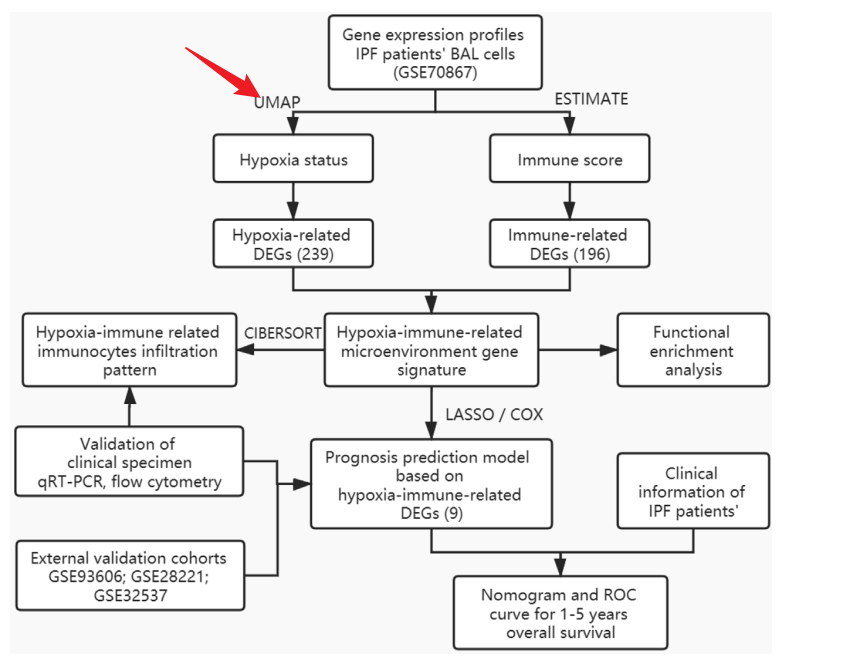
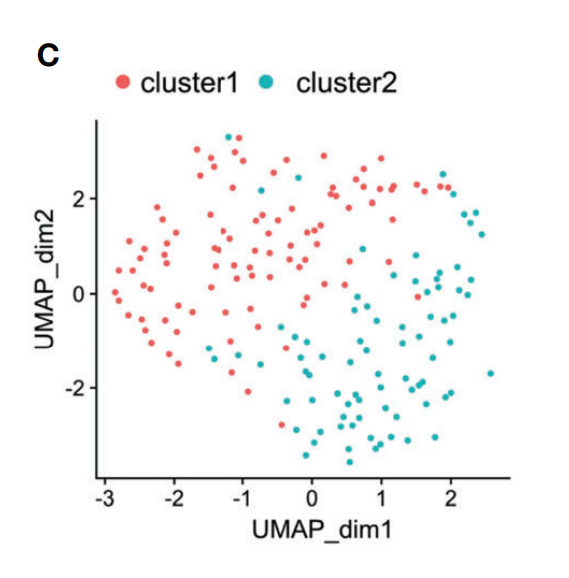
本篇推文所在分析流程位置如图。对应图为figure2C。
示例数据和代码领取
点赞、在看本文,分享至朋友圈集赞20个并保留30分钟,截图发至微信mzbj0002领取。
木舟笔记2022年度VIP可免费领取。
木舟笔记2022年度VIP企划
权益:
2022年度木舟笔记**所有推文示例数据及代码(在VIP群里实时更新)**。

木舟笔记科研交流群。
半价购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
收费:
99¥/人。可添加微信:mzbj0002 转账,或直接在文末打赏。

方法简析

首先作者查找了MSigDB数据库中的缺氧基因集,并在之前去出国批次效应后合并的表达矩阵中提取改基因集的表达矩阵。
然后接下来就出现了本文的第一个存在疑问的地方。众所周知,umap是一种常用的降维算法,并没有聚类的作用,所以作者这里应该漏写了聚类的具体方法。基于此,又有聚类后的降维可视化,或者基于降维数据的聚类可视化。基于作者对umap的大量着墨,我这里就利用k-means算法对umap结果进行聚类并可视化。
开始分析
最后
以上就是耍酷星星最近收集整理的关于Front Immunol 复现 | 2. 一个基于缺氧基因集的数据降维聚类分组方法(umap,MSigDB)的全部内容,更多相关Front内容请搜索靠谱客的其他文章。








发表评论 取消回复